- 07 octobre 2024
- | Source: Foodprocess
Le séquençage du génome entier des bactéries à portée de main
Le matériel portable de séquençage de l'ADN en temps réel met le séquençage du génome entier des bactéries à la portée des entreprises agroalimentaires. Mais quelle est la qualité et la fiabilité des séquences d'ADN et dans quelle mesure peut-on les utiliser pour identifier les bactéries? Le projet SIPORE a permis de répondre à cette question pour Listeria monocytogenes et plusieurs sérovars de Salmonella.
Grâce aux progrès rapides des nanotechnologies et de la bio-informatique, il est désormais possible de lire le génome d'une bactérie en moins d'une journée. Le séquençage du génome entier est ainsi devenu une méthode pratique pour identifier rapidement les bactéries individuelles. En outre, les données du génome permettent de déduire beaucoup de choses sur les propriétés spécifiques d'une bactérie isolée. C'est pourquoi il est intéressant pour les entreprises alimentaires d'intégrer cette technologie dans leurs contrôles de qualité ou dans le développement de leurs produits (en particulier pour les aliments fermentés).
Avec l'apparition d'équipements portables de séquençage 'nanopore', le séquençage du génome entier est de plus en plus à la portée des entreprises alimentaires. Cependant, dans cette technologie rapide, des erreurs se glissent parfois. Les bactéries peuvent-elles donc être identifiées de manière fiable jusqu'au niveau de la souche? Cela peut être important si l'on veut utiliser cette technologie pour découvrir les voies et les sources de contamination dans un environnement de production, par exemple. Pour en avoir le cœur net, le projet SIPORE a étudié les possibilités offertes par deux agents pathogènes clés d'origine alimentaire: Listeria monocytogenes et Salmonella (différents sérovars).
Analyse de séquences d'ADN par nanopore
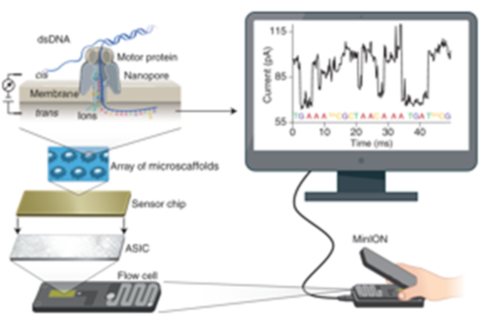
L'analyse des séquences d'ADN est le processus de détermination de la séquence de nucléotides d'un brin d'ADN. L'analyse de séquence par nanopore est une technologie développée il y a une dizaine d'années qui permet l'analyse directe et en temps réel de longs fragments d'ADN (> 10 000 nucléotides). L'ADN monocaténaire passe ainsi à travers de petits pores (appelés 'nanopores'). Les brins d'ADN qui passent provoquent de petites variations dans le courant électrique qui traverse le nanopore. En mesurant ces variations, on détermine la séquence de nucléotides (figure 1). Grâce à des algorithmes bioinformatiques, les séquences d'ADN des fragments sont assemblées en une seule séquence génomique.
De la bactérie à la séquence du génome entier
La mise en œuvre pratique de l'analyse de séquences d'ADN par nanopores comprend deux parties: les travaux pratiques en laboratoire (de la préparation de l'échantillon au démarrage de l'analyse de la séquence d'ADN) et l'analyse des données à l'aide de la bio-informatique. Pour la première partie, un protocole a été optimisé dans SIPORE qui comprend les étapes suivantes: extraction de l'ADN (à partir de bactéries isolées), réparation de l'extrémité de l'ADN, ligature et nettoyage de l'adaptateur et chargement de la cellule d'écoulement.
Pour la deuxième partie, des 'pipelines' ont été développés dans lesquels différents algorithmes ont été testés pour les différentes étapes afin d'obtenir une séquence du génome entier. Des protocoles personnalisés permettant d'analyser jusqu'à 20 échantillons simultanément ont également été testés. Il est apparu clairement qu'il fallait trouver un compromis entre l'efficacité (le nombre d'échantillons à analyser simultanément) et la fiabilité recherchée.
De la séquence du génome entier à l'identification et plus encore...
Les séquences génomiques obtenues ont été comparées à des bases de données. Par exemple, pour Listeria monocytogenes, il existe des bases de données très détaillées au niveau des souches (cgMLST ou 'core genome multilocus sequence typing'). Il s'est avéré possible d'identifier les bactéries Listeria monocytogenes jusqu'au niveau cgMLST grâce aux protocoles et au 'pipeline' mis au point.
Il a été possible d'aller encore plus loin dans la caractérisation. En utilisant des cellules de flux pour l'analyse des brins complémentaires et des nanopores plus longs pour la double lecture des séquences, il a été possible de distinguer des isolats dont les génomes ne différaient que de quelques nucléotides. Pour Salmonella, un 'pipeline' a été mis au point qui a permis l'identification de 12 sérovars.

Dans la séquence génomique obtenue, certains algorithmes ont permis de rechercher des gènes intéressants, tels que ceux impliqués dans la sensibilité aux désinfectants, la formation de biofilms, les réactions au stress, la virulence et la résistance aux antibiotiques, entre autres. De nombreuses informations ont été obtenues sur les propriétés des bactéries isolées.
Ces résultats montrent le potentiel du séquençage 'nanopore' pour la réalisation d'analyses microbiennes. Pour mettre cette technologie à la portée des entreprises alimentaires, il est important de développer des protocoles et des 'pipelines' spécifiques avec une bio-informatique adaptée à l'organisme cible visé (dans le projet, il s'agissait de souches de Listeria monocytogenes et de sérovars de Salmonella). Il convient toutefois de noter que les résultats du projet ont été obtenus par l'isolement de l'ADN de bactéries isolées.
D'autres développements sont nécessaires pour analyser également des échantillons plus complexes, qui contiennent de l'ADN d'autres organismes en plus de l'organisme cible. Pour les applications biomédicales, des recherches prometteuses sont déjà en cours sur l'approche de l'échantillonnage adaptatif qui permet, par exemple, au séquençage par nanopores d'identifier l'ADN peu fréquent d'agents pathogènes dans des échantillons de sang de patients (ADN de l'hôte commun). Les projets LeapSEQ et MetaTec sont des exemples d'applications du séquençage par nanopore pour les analyses métagénomiques.
Inspirés par ces projets, Howest et l'ILVO, à la suite du projet SIPORE, ont déjà mené avec succès un premier essai de détection de Listeria monocytogenes dans un mélange microbien plus complexe par le biais d'un échantillonnage adaptatif. Ce type de recherche pourrait conduire à l'application du séquençage 'nanopore' à des échantillons agroalimentaires complexes dans un avenir relativement proche.
Après avoir lu cet article, êtes-vous intéressé par l'application du séquençage portable de l'ADN en temps réel dans votre exploitation ou voyez-vous des possibilités d'application intéressantes qui nécessitent davantage de recherche et/ou de développement? Si c'est le cas, veuillez contacter steven.vancampenhout@flandersfood.com pour plus d'informations.
